Un prion qui n’est plus sans gène
Pourquoi certaines erreurs dans la lecture du code génétique sont-elles conservées au cours de l’évolution ? Quel est leur rôle dans l’expression de certains gènes ? En se penchant sur ces questions, des chercheurs de l’équipe de génétique moléculaire à l’Institut de Génétique et Microbiologie viennent d’établir un lien entre une protéine produite par une de ces lectures alternatives et un des prions de la levure Saccharomyces cerevisiae.
Le code génétique est un système de correspondance permettant de traduire une séquence d’acides nucléiques (ADN et ARN) en protéine. La première étape de l’expression des gènes est la transcription qui permet d’exprimer l’information contenue dans l’ADN en une copie ARN. Cet ARN messager transmet l’information génétique jusqu’aux ribosomes où s’effectue la traduction, deuxième étape de l’expression des gènes. Des différents niveaux du transfert de l’information génétique, la traduction est à l’heure actuelle le moins bien connu. Au sein de l’Institut de Génétique et Microbiologie, notre équipe s’intéresse aux erreurs de traduction et à leur incidence sur l’expression génétique.
Le mécanisme de la traduction est l’œuvre des ribosomes, les usines productrices de protéines. Tous les organismes vivants utilisent les ribosomes pour interpréter le code génétique porté par les ARN messagers (ARNm) et synthétiser les protéines, c’est la traduction génétique. Ainsi à chaque triplet de bases (codon) correspond un ARN de transfert (ARNt) portant l’acide aminé correspondant au codon lu. Lorsque le ribosome rencontre le codon d’initiation, la traduction commence. Elle s’arrête lorsque le ribosome rencontre un codon stop. En réalité, cet arrêt est provoqué par deux protéines qui vont pénétrer dans le ribosome. Appelées facteurs de terminaison, leur rôle est essentiel pour achever la synthèse des protéines ; elles vont reconnaître sur l’ARN messager le codon « stop », permettant ainsi la libération de la protéine synthétisée.
Erreurs de traduction
Il est essentiel que l’information génétique soit correctement décodée par le ribosome, sinon la protéine synthétisée ne possèdera pas les caractéristiques biochimiques attendues ce qui pourra avoir des conséquences parfois graves pour la cellule. Deux types d’erreurs peuvent se produire au moment de la traduction. Les erreurs spontanées qui se produisent de façon aléatoire sur tous les gènes mais à un très faible niveau. Elles sont dues à l’absence de ponctuation du code génétique. En effet, une fois que la lecture a démarré, si à un moment donné le ribosome change de cadre de lecture en glissant d’une base, il n’aura aucun moyen de revenir dans la bonne phase de lecture, la protéine synthétisée ne sera pas fonctionnelle. Ces erreurs sont extrêmement rares, on estime leur fréquence inférieure à 5×10-5 par codon. Le deuxième type d’erreurs concernent des erreurs dites programmées (parce qu’elles sont provoquées par des motifs présents sur l’ARNm), on parle aussi de recodage. Contrairement aux erreurs spontanées, les erreurs programmées ne concernent que certains gènes mais au lieu d’empêcher l’expression de la protéine, elles vont permettre l’expression d’une protéine possédant de nouvelles caractéristiques. Notre équipe s’intéresse à ces erreurs programmées pour comprendre le rôle qu’elles jouent dans la régulation de l’expression de certains gènes, en particulier chez la levure S. cerevisiae.
Un des décalages programmés du cadre de lecture en +1 les mieux conservés au cours de l’évolution, est celui que l’on retrouve dans le gène OAZ codant l’antizyme (une petite protéine régulant négativement la biosynthèse des polyamines) (figure). Lors de sa lecture, le ribosome change de cadre de lecture à un endroit précis et poursuit la traduction jusqu’au codon stop suivant permettant la synthèse de la protéine fonctionnelle.
Lien entre le prion [PSI+] et les polyamines
Nous nous sommes intéressés à eRF3, un des deux facteurs de terminaison. Il se trouve que cette protéine peut changer de conformation et s’agréger. Cette agrégation provoque l’inactivation partielle du facteur de terminaison. Cette nouvelle conformation étant capable de convertir les protéines eRF3 nouvellement synthétisées et de se transmettre aux cellules filles, on parle alors du prion [PSI+]. L’apparition de [PSI+] dans une cellule s’accompagne d’un grand nombre de modifications, aussi bien morphologiques que métaboliques (résistance aux stress). Malgré de nombreux travaux, l’origine de ces modifications est encore mal comprise, en particulier aucun gène n’était connu pour être directement régulé par ce prion.
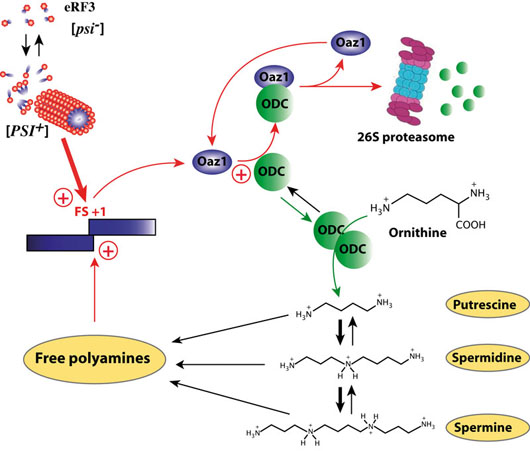
Figure: voie de régulation de la biosynthèse des polyamines. OAZ= antizyme; ODC= Ornithine décarboxylase.
Les résultats que nous avons obtenus montrent que le prion [PSI+] stimule fortement le décalage du cadre de lecture du gène OAZ (l’antizyme de l’ornithine décarboxylase) et permet ainsi d’augmenter la quantité d’antizyme dans la levure. Or l’antizyme contrôle négativement toute la voie de biosynthèse des polyamines (qui sont de petites molécules chargées positivement impliquées dans de très nombreuses fonctions physiologiques dans la cellule). L’augmentation de la synthèse de l’antizyme entraine une diminution de la concentration intracellulaire de polyamines. Nous avons montré que cette baisse permet d’expliquer la moitié des nombreux phénotypes associés au prion [PSI+]. OAZ est donc le premier gène cellulaire dont l’expression est directement contrôlée par le prion [PSI+].
Olivier Namy
Contact
Dr. Olivier NAMY
Equipe Génétique Moléculaire de la Traduction
Institut de Génétique et Microbiologie, bât. 400
Université Paris-Sud 11
Tél : 01 69 15 46 34
olivier.namy@u-psud.fr
http://www.igmors.u-psud.fr/rousset/namy/index.fr.html
Références
Epigenetic control of polyamines by the prion [PSI+].
Namy O, Galopier A, Martini C, Matsufuji S, Fabret C, Rousset JP.
Nat Cell Biol. 2008 Sep;10(9):1069-75.